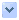從達爾文以降,演化生物學家一直想要建立地球上各種生物的生命之樹。分子演化學家所選用的方法大多是比較各種不同生物的幾個基因之序列,來為它們建立親緣關係。可是今演化學家頭痛的是,選用不同的基因往往會畫出不一致的演化樹。而今這個難題終於可望利用基因組的資料來解決。
威斯康辛大學和霍華休斯醫學研究院的Sean B. Carroll等人證實基因組的資料可以協助繪出解析度前所未有地清晰的演化樹。這個發現的重要性在於,一個生命之樹的精確預測,是生物學家對地球數十億年的演化史之瞭解,是非常關鍵的。對地球生命史精確的瞭解,不僅能滿足演化生物學家的好奇心,對新藥開發、作物改良、新突現疾病的研究和生態保育都是非常重要的。
為了得到可信度高的生命之樹,科學家從八○年代開始就利用基因序列來推斷各種生物的演化歷史。基因帶有生物的遺傳訊息碼,不會受到生物個體後天生長情況的影響,比起外在特徵更可靠。透過幾個基因的比較,科學家就能安排出不同動植物和微生物之間的親緣關係。可是這個方法是問題是利用單一基因繪出的演化樹往往缺乏足夠的可信度。每個基因都有不同的故事,因為都經過了不同的選擇壓力,於是它們帶有的訊息包含了選擇的結果和生命的歷史,於是每個基因都有自己的偏見。所以不同的基因就能做出不同的演化樹,各種不同基因簡直就是在百家爭鳴。
當然多用些基因會解決部分的問題,而Carroll等人就在要探討到底要用到多少基因才足夠。利用八種酵母菌的基因組資料,他們從中選用了上百個同源基因,而結果繪出的演化樹無疑就是獨一無二的。儘管每個不同基因有自己的故事要說,可是把它們都集合起來之後,它們就能完精確地說出一個生命演化史。
這個方法讓科學家終於能夠建立更建全的生命之樹。拜科技進步所賜,科學家現在終於有了各種不同生物的基因組資料,從今以後親緣關係樹的建立,也將邁入一個新的層次。
原學術論文:
Rokas, A., Williams, B. L., King, N. & Carroll, S. B. Genome-scale approaches to resolving incongruence in molecular phylogenies. Nature 425, 798 - 804 (23 October 2003); doi:10.1038/nature02053
--參考來源:
EurekAlert! (10/22/2003) -- New genomic data helps resolve biology's tree of life
http://www.eurekalert.org/pub_releases/2003-10/uow-ngd101703.php
Gee, H. Evolution: Ending incongruence. Nature 425, 782 (23 October 2003); doi:10.1038/425782a
http://www.nature.com/cgi-taf/DynaPage.taf?file=/nature/journal/v425/n6960/full/425782a_fs.html
--相關連結:
A Tree of Life
http://intl.sciencemag.org/feature/data/tol/
Howard Hughes Medical Institute
http://www.hhmi.org/
University of Wisconsin-Madison
http://www.wisc.edu/
注意圖中他們的bootstrap值都是100/100/100(ML on nucleotides/MP on nucleotides/MP on amino acids)哦!Bootstrap是一種重取樣檢驗法,它代表演化樹的可信度,利用三種建構演化樹的方法,其重取樣一千次建構出的演化樹還是百分之百地和原先建構的演化樹完全一樣!摘自原學術論文。
本文原刊登於SciScape:
http://www.sciscape.org/news_detail.php?news_id=1297