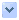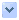四、 魚類mitogenome序列研究
(一)魚類完整mitogenome研究之發展
魚類粒線體DNA之完整序列研究最早源於1992年,Tzeng等學者欲研究產於山區的一種台灣纓口鰍Crossostoma lacustre之mitogenome的結構與組成,遂以轉殖(cloning)的方式,先利用限制酶HindIII將卵細胞中之mtDNA切割成9個片段,再轉殖入質體並分別以不同限制酶作標定,完全定序(complete sequencing)全長共16,558 bp的mitogenome,其中包含D-loop基因、2個rRNA基因、22個tRNA基因及13個蛋白質基因(Fig. 18),發現其基因組成的種類與排列方式都與其他脊椎動物相似,且缺乏intron(Table 4)(Tzeng et al., 1992;Lee et al., 2001)。接著陸續有十餘種魚種之mtDNA被完全定序,包括鱈魚、海鰻、角鯊、鱒魚、肺魚、鯊魚、金魚、冰魚及腔棘魚等(Hurst et al., 1999)。
而2000年Inoue等學者為了研究日本產量最多的沙丁魚Sardinops melanostictus之mtDNA,嘗試利用長鏈PCR(long-PCR)搭配30組小片段序列之primer進行PCR增幅(Fig. 19),合併應用的巢式PCR(nested PCR)技術,成功的在未經轉殖的情況下定出了此魚種的mtDNA完整基因體為16,881 bp;而之後亦陸續有學者以PCR增幅的方法取代傳統的轉殖,來作魚類mitogenome完整序列的定序及分析(Lee et al., 2001;Inoue et al., 2001a;Inoue et al., 2001b;Delarbre et al., 2002)。
(二)序列組成分析
為了與親源相近的彩虹鱒魚(rainbow trout)mtDNA比對差異性,Hurst等(1999)訂定出大西洋鮭魚(Atlantic salmon;Salmo salar)mtDNA的完整基因體,長度為16,665 bp,基因組成其他硬骨魚綱魚種相同。將其13個蛋白密碼基因與彩虹鱒魚相比對,發覺相似度為84.3~95.0%,13個蛋白密碼轉譯成蛋白的相似度更高達93.9~100%(Table 5);此外Hurst等並建構出OL上的stem-loop構形,發現鮭魚的組成(stem/12 bp;loop/13 nt)較接近人類(stem/11 bp; loop/12 nt)而非鱒魚(stem/11 bp;loop/17 nt)(Fig. 20),此外亦與沙丁魚(stem/12 bp;loop/12 nt)相似(Inoue et al., 2000)。
Tzeng等(1992)將最早完成定序的魚種C. lacustre,與其他物種相比較整個mtDNA上H-strand的四組鹼基組成,發現其與鯉魚、鯨魚相同,皆為A>C>T>G;而雞和人類為C>A>T>G,其他物種包括牛、鼠、爪蟾、果蠅與海膽等則為A>T>C>G(Table 6)。此外在C/G比例的部分其值為1.69,大致與變溫動物爪蟾(1.64)、鯉魚(1.74)相似;低於恆溫動物的牛(1.92)、人類(2.38),而高於非脊椎動物的海膽(1.23)與果蠅(1.33)。就整個mtDNA的總長度而言,魚類(泥鰍、鯉魚)與哺乳類、鳥類等脊椎動物的總長度差別不大(約在16.3~16.7 kb間),但卻明顯比蛙類的爪蟾少了1 kb左右;其他各基因的比較,差異最大的D-loop區,除了海膽特別短只有121 bp、爪蟾特別長高達2134 bp外,其他哺乳動物與魚類無明顯差異,大致為1 kb左右(Table 7)(Tzeng et al., 1992)。Elmerot等(2002)在大致比對70種魚類mitogenome的長度後(Table 8),發覺少有低於16.1 kb的情形;而分析其序列短缺的地點,大多發生於control region區,不會影響功能性基因的表現。
(三)系統演化分析
以mtDNA上的部分序列片段作為親緣演化的研究很多,然而其呈現的結果,皆不如以整個mitogenome為依據的分析詳盡(Miya et al., 2000)。Inoue等曾就傳統硬骨魚分類的5組各2種代表魚種,與另外2個軟骨魚種(Fig. 21),以整個mitogenome中連續的13個蛋白基因及22個tRNA基因,經由統計軟體進行系統演化樹分析(Fig. 22),結果顯示其分類與傳統的演化分類相同,且可依鹼基置換的比例,更詳細的區別演化分支與親緣關係(Inoue et al., 2001a)。
2002年Elmerot等將虎河魨Fugu rubripes之mitogenome完整定序(16,446 bp)後,進入基因庫搜尋所有魚類,查詢出有29目(order)共69物種(species)曾建立完整的mitogenome序列資料;學者進一步將這些不同魚種的基因體,依其轉譯成的胺基酸序列,以軟體分析重建其模擬的系統親緣關係(phylogenetic relationship)(Fig. 23),學者分析大部分魚種的演化進度差異不大,只有少數魚種在演化過程中有加速(accelerate)的情形。整體而言,以mitogenome為基礎建構而成的演化樹,與傳統的型態學分類一致,且更能詳細推測演化進度,在物種親緣關係的研究上可作為參考的重要依據(Miya et al., 2000;Inoue et al., 2001a;Elmerot et al., 2002)。
(四)特殊基因型態
Delabre等(2002)研究一種八目類鰻魚(hagfish)布氏粘盲鰻Eptatretus burgeri的完整mitogenome,定出全長17,168 bp的mtDNA序列,發現這種古老的圓口類(cyclostomes)生物(即無頷類生物;脊椎動物分有頷gnathostomes及無頷兩大類)之mtDNA基因型態與排列順序,大致與其他魚類相同;然而其control region則特別長,達1,858 bp(Table 9),其中5’端的257 bp序列重複出現兩次,第三次重複則只有256 bp(少了一個鹼基T),第四次則只有部分134 bp出現重複的情形;此與其他脊椎類動物1,000 bp左右的control region長度相比有明顯差異,Delabre等認為此可作為這個現存的活化石物種在演化上與其他魚種相異的標定特徵。
Inoue等(2001b)將一海鰻類魚種,星康吉鰻(Conger myriaster)的mitogenome定出總長為18,705 bp的完整序列,較其他脊椎動物長約2 kb,進而分析序列組成順序,發現其獨有的基因型態;在C. myriaster之mitogenome中的ND6、tRNAGlu與tRNAPro等3組基因位於control region與tRNAPhe間,而非傳統脊椎動物的ND5與control region間,且多出一段未編碼(non-encode)的片段(Fig. 24),推測其為演化上rearangement多重變異所造成的結果。目前在四種鳥類的mitogenome中亦曾發現類似的基因型態,不過其為ND6、tRNAGlu與tRNAPro等3組基因完全自mitogenome刪除,與C. myriaster的情況不盡相同。
為了研究一種自體授精(self-fertilize)將魚Rivulus marmoratus的mitogenome,Lee等(2001)亦以巢式PCR技術定序出全長為17,329 bp的完整mtDNA序列,然而經由比對後發現R. marmoratus有2個control region(Fig. 25)。其中control region I所在位置與其他魚類相同,於cyt b與12S rRNA兩基因之間;而control region II則存在於16S rRNA旁的tRNALeu與ND1兩基因之間(Table 10)。Control region I長度為887 bp,而control region II則為795 bp,兩者相似度頗高,中間一段735 bp長的區域幾乎完全相同,只有兩個nucleotide的差異(Fig. 26),推論control region II應該源自於control region I。而為了解此基因型態是否廣泛存在於R. marmoratus物種中,學者亦針對不同個體及卵細胞,以及Rivulidae屬魚類其他魚種作電泳測試,證實此特殊的基因型態在魚類中僅R. marmoratus獨有(Fig. 27);然而比對其他脊椎動物,則發現在已定出完整mtDNA序列的2種蛇類、鸚鵡及壁虱,皆曾發現有類似的基因型態(Lee et al., 2001)。
五、 結論
1. mitogenome由13個mRNA基因、22個tRNA基因、2個rRNA基因及control region所組成;而魚類mitogenome的序列大小、基因組成皆與其他脊椎類動物相似。
2. 除了某些二枚貝外,動物界所有物種的mitogenome皆為單向的母系遺傳,沒有基因重組的情形。
3. mitogenome序列重排及鹼基置換的發生率較高,因而變異度在演化上容易產生分歧;而以mitogenome為基礎建構而成的演化樹,較傳統的型態學分類更能詳細推測演化進度。
4. 在魚類mitogenome發現特殊的基因型態,大多發生於未紀錄基因密碼的control region區。
Reference
Bogenhagen, D. F. and Clayton, D. A. 2003. The mitochondrial DNA replication bubble has not burst. Trands in Biochemical Sciences 28 (7): 357-360.
Bremer, J. R. A., Naseri, I. and Ely, B. 1997. Orthodox and unorthodox phylogenic relationships among tunas revealed by the nucleotide sequence analysis of the mitochondrial DNA control region. Journal of Fish Biology 50: 540-554.
Bromham, L., Eyre-Walker, A., Smith, N. H. and Smith, J. M. 2003. Mitochondrial Steve: paternal inheritance of mitochondrial in humans. Trends in Ecology and Evolution 18 (1): 2-4.
Broore, J. L. and Brown, W. M. 1998. Big trees from little genomes: mitochondrial gene as a phylogenic tool. Current Opinion in Genetics & Development 8: 668-674.
Chinnery, P. F. and Turnbull, D. M. 2000. Mitochondrial DNA mutations in the pathogenesis of human disease. Molecular Medicine Today 6: 425-432.
Clayton, A.D. 2000. Vertebrate mitochondrial DNA—A circle of surprises. Experimental Cell Research 255: 4-9.
Curole, J. P. and Kocher, T. D. 1999. Mitogenomes: digging deeper with complete mitochondrial genomes. Trends in Ecology & Evolution 14 (10): 394-398.
Delarbre, C., Gallut, C., Barriel, V., Janvier, P. and Gachelin, G. 2002. Complete mitochondrial DNA of the hagfish, Eptatretus burgeri: the comparative analysis of mitochondrial DNA sequences strongly supports the cyclostome monophyly. Molecular Phylogenetics and Evolution 22 (2): 184-192.
Elmerot, C., Arnason, U., Gojobori, T. and Janke, A. 2002. The mitochondrial genome of the pufferfish, Fugu rubripes, and ordinal teleostean relationships. Gene 295: 163-172.
Garesse, R. and Vallejo, C. G. 2001. Animal mitochondrial biogenesis and function: a regulatory cross-talk between two genome. Gene 263: 1-16.
Hurst, C. D., Bartlett, S. E., Davidson, W. S. and Bruce, I. J. 1999. The complete mitochondrial DNA sequence of the Atlantic salmon, Salmo salar. Gene 239: 237-242.
Inoue, J. G., Miya, M., Tsukamoto, K. and Nishida, M. 2000. Complete mitochondrial DNA sequence of the Japanese sardine Sardinops melanostictus. Fisheries Science 66: 924-932.
Inoue, J. G., Miya, M., Tsukamoto, K. and Nishida, M. 2001a. A mitogenomic perspective on the basal teleostean phylogeny: resolving higher-level relationships with longer DNA sequences. Molecular Phylogenetics and Evolution 20 (2): 275-285.
Inoue, J. G., Miya, M., Tsukamoto, K. and Nishida, M. 2001b. Complete mitochondrial DNA sequence of Conger myriaster (Teleostei: Anguilliformes): novel gene order for vertebrate mitochondrial genomes and the phylogenetic implications for anguilliform families. Journal of Molecular Evolution 52 (4): 311-320.
Lee, J. S., Miya, M., Lee, Y. S., Kim, C. G., Park, E. H., Aoki, Y. and Nishida, M. 2001. The complete DNA sequence of the mitochondrial genome of the self-fertilizing fish Rivulus marmoratus (Cyprinodontiformes, Rivulidae) and the first description of duplication of a control region in fish. Gene 280: 1-7.
Leonard, J. V. and Schapira, A. H. V. 2000. Mitochondrial respiratory chain disorders I: mitochondrial DNA defects. The Lancet 355: 299-304.
Mandavilli, B. S., Santos, J. H. and Houten, B. V. 2002. Mitochondrial DNA repair and agong. Mutation Research 509: 127-151.
Miya, M. and Nishida, M. 2000. Use of mitogenomic information in teleostean molecular phylogenetics: a tree-based exploration under the maximum-parsimony optimality criterion. Phylogenetics and Evolution 17 (3): 437-455.
Rokas, A., Ladoukakis, E. and Zouros, E. 2003. Animal mitochondrial DNA recombination revisited. Trends in Ecology and Evolution 18 (8): 411-417.
Schon, E. A. 2000. Mitochondrial genetics and disease. Trends in Biochemical Sciences 25 (11): 555-560
Stoneking, M. and Soodyall, H. 1996. Human evolution and the mitochondrial genome. Current Opinion in Genetics & Development 6: 731-736.
Taanman, J.-W. 1999. The mitochondrial genome: structure, transcription, translation and replication. Biochimica et Biophysica Acta 1410: 103-123.
Tamura, K. 2000. On the estimation of the rate of nucleotide substitution for the control region of human mitochondrial DNA. Gene 259: 189-197.
Tzeng, C. S., Hui, C. F., Shen, S. C. and Huang, P. C. 1992. The complete nucleotide sequence of the Crossostoma lacustre mitochondrial genome: Conservation and variations among vertebrates. Nucleic Acids Research 20 (18): 4853-4858.
文章定位: